2/3 des amorces PCR publiées n'ont pas une efficacité optimale pour la détection !
Avec les récentes avancées en biologie, les méthodes de détection traditionnelles (cultures cellulaires, tests biochimiques, sérotypage) sont progressivement en train d'être remplacées par les technologies basées sur l'ADN (PCR, qPCR, MLVA, MLST, NGS, etc.). Ces méthodes de biologie moléculaire ont les avantages d'être plus rapides (résultats en moins d'une journée dans certains cas), plus robustes (on évite par exemple le biais de fitness vis à vis du milieu de culture) et apportent un niveau d'informations plus important.
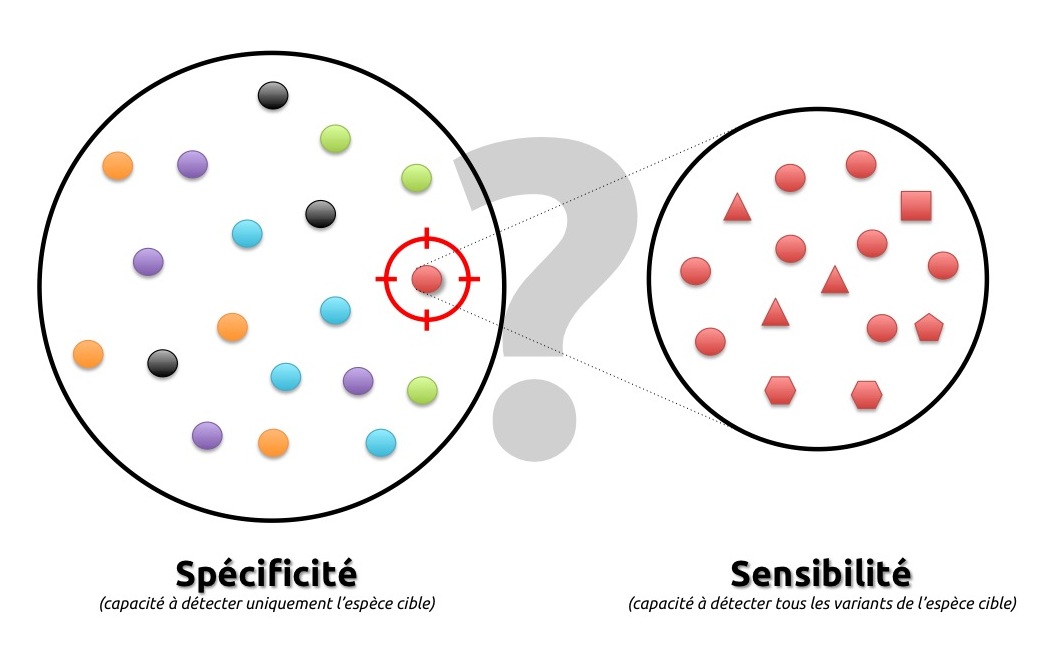
La majorité de ces techniques reposent sur l'amplification du matériel génétique par PCR. Néanmoins cette étape est tributaire de la qualité des amorces ou primers, éléments chimiques responsables du ciblage de l'ADN. Au cours de nos recherches pour la Délégation Générale de l'Armement, nous avons montré que deux tiers des amorces PCR publiées dans la littérature scientifique et servant à détecter les gènes de pathogénicité de Vibrio cholerae n'ont pas une efficacité optimale.
A la suite de ces constats, nous avons développé BiomandaDetect, une méthode bioinformatique unique permettant de vérifier, améliorer ou concevoir des amorces de PCR. Ses atouts :
En fonction de vos besoins, ce service peut être couplé avec une étape de data-mining (fouille de données) pour rassembler un maximum d'éléments nécessaires à un développement optimal de votre solution de détection (phylogénie, origine et date de prélèvement, nom de la souche, serovar ou serotype, hôte, etc.). Nous pouvons intervenir sur n'importe quel type d'organisme vivant (virus, bactéries, moisissures/champignons, parasites, autres protistes, métazoaires, plantes et archaea) et à n'importe quel niveau taxonomique (genre, espèce, génotype, etc.).